Wissenschaftlern der Universität Bonn und des Forschungszentrums caesar ist es gelungen, Proteine nach einer exakt definierten Zeitspanne ultraschnell einzufrieren. Die Moleküle konnten sie dann auf Bruchteile eines Millionstel Millimeters genau vermessen. Die Methode ermöglicht es, schnelle Strukturänderungen in Enzymen und Nukleinsäuren zeitlich zu verfolgen. Die Ergebnisse erscheinen im Journal of the American Chemical Society.
Wer wissen möchte, wie die räumliche Struktur eines bestimmten Biomoleküls aussieht, dem steht dazu ein ganzes Arsenal von Werkzeugen zur Verfügung. Zu den bekanntesten zählen die Elektronenmikroskopie und die Röntgenbeugung, die selbst kleinste Details eines Proteins sichtbar machen können. Das Problem dabei: Diese Methoden liefern in der Regel statische Bilder. Ein langfristiges Ziel vieler Forschungsgruppen weltweit ist es daher, die Bewegungen eines Zellmoleküls wie in einem Film zeitlich zu verfolgen, während es seine Arbeit ausführt. Die Arbeitsgruppen um Prof. Dr. Olav Schiemann vom Institut für Physikalische und Theoretische Chemie der Universität Bonn und Prof. Dr. Benjamin Kaupp vom Forschungszentrum caesar der Max-Planck-Gesellschaft sind diesem Ziel nun ein Stück nähergekommen.
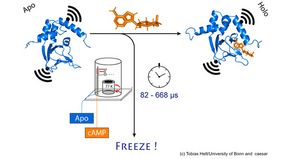
Als Untersuchungsobjekt diente ihnen ein Ionenkanal. Dabei handelt es sich um eine mikroskopisch kleine Pore in der Zellmembran, die für Ionen - also geladene Teilchen - durchlässig ist. „Der von uns untersuchte Kanal ist normalerweise geschlossen“, erklärt Schiemann. „Erst wenn ein zellulärer Botenstoff namens cAMP an ihn bindet, öffnet er sich. Wir wollen wissen, wie dieser Vorgang genau abläuft.“
Mini-Magneten zur Abstandsmessung
Dazu mischten die Wissenschaftler zunächst das Kanalprotein und cAMP und froren die Lösung dann ein. Im gefrorenen Zustand konnten sie das Protein nun räumlich vermessen. Sie hatten dazu an zwei Stellen des Kanals molekulare Elektromagneten befestigt. Den Abstand dieser Magneten kann man mit einer raffinierten Methode, genannt PELDOR, bis auf wenige hundert Milliardstel Millimeter genau bestimmen, wie mit einem molekularen Zollstock. Die Methode wurde in den vergangenen Jahren in Schiemanns Arbeitsgruppe entscheidend weiterentwickelt und verbessert.
„Allerdings erhalten wir so nur ein Standbild der cAMP-Bindung an den Ionenkanal“, sagt Schiemann. „Wir haben den Einfriervorgang daher zu verschiedenen Zeiten nach der Mischung beider Moleküle wiederholt. Auf diese Weise konnten wir die Bewegungen im Protein nach der cAMP-Bindung rekonstruieren - wie bei einem Film, der ja auch aus einer Abfolge von Bildern besteht.“
Kern des Verfahrens ist eine ausgeklügelte Methode, die es erlaubt, Proben sehr schnell und zu einem präzisen Zeitpunkt einzufrieren. Die Technik namens „microsecond hyperquench freezing“ (abgekürzt MHQ) stammt ursprünglich von der Universität Delft, geriet aber in Vergessenheit. Sie wurde von Kaupps Arbeitsgruppe wiederentdeckt und entscheidend weiterentwickelt.
„Bei der MHQ werden das cAMP-Signalmolekül und der Ionenkanal zunächst ultraschnell miteinander gemischt“, erklärt Kaupp. „Dann wird die Mischung in einem haarfeinen Strahl auf einen sehr kalten Metallzylinder bei -190 Grad Celsius geschossen, der sich mit 7.000 Umdrehungen pro Minute dreht. Besonders knifflig war es, die Proben für die PELDOR Messung im gefrorenen Zustand von der Metallplatte in dünne Glasröhrchen zu überführen. Dafür mussten wir Spezialwerkzeuge entwickeln und bauen.“
Tiefkühlung in 82 Millionstel Sekunden
Insgesamt dauert der gesamte Misch- und Einfriervorgang nur 82 Millionstel Sekunden. „Wir können dadurch sehr schnelle Änderungen in der räumlichen Struktur von Proteinen sichtbar machen“, erläutert Tobias Hett, einer der beiden Doktoranden, die maßgeblich zum Erfolg beigetragen haben. Vorteil des Verfahrens ist seine Kombination aus hoher räumlicher und zeitlicher Auflösung. „Für die Messung dynamischer Vorgänge im Reich der Moleküle ist das ein großer Fortschritt“, betont Kaupp.
Die Wissenschaftler wollen nun auf diese Weise weitere Biomoleküle unter die Lupe nehmen. Sie erhoffen sich neue Erkenntnisse, etwa zur Funktionsweise bestimmter Enzyme und Nukleinsäuren. Wie wichtig solche Einblicke sind, zeigen beispielsweise die weltweiten Forschungs-Arbeiten zum SARS-Coronavirus-2: Das sogenannte Spike-Protein des Virus durchläuft bei der Infektion von menschlichen Zellen ebenfalls eine Strukturänderung. Die Aufklärung dieses Mechanismus hat unter anderem Hinweise auf mögliche Angriffspunkte für neue Medikamente geliefert.
Die Herstellung der Proben, ihre Vermessung und die Analyse der Messsignale ist sehr komplex. Die Ergebnisse der Studie sind daher auch Ausdruck einer erfolgreichen wissenschaftlichen Kooperation mit Wissenschaftlern um Prof. Dr. Helmut Grubmüller vom Max-Planck-Institut für biophysikalische Chemie in Göttingen und Prof. Dr. Heinz-Jürgen Steinhoff von der Universität Osnabrück.
Beteiligte Institutionen und Förderung:
An der Studie waren neben dem Forschungszentrum caesar und der Universität Bonn die Universität Osnabrück und das Max-Planck-Institut für biophysikalische Chemie in Göttingen beteiligt. Die Arbeiten werden von der Deutschen Forschungsgemeinschaft (DFG) im Rahmen des Koselleck-Programms gefördert und aus Mitteln des Transdisziplinären Forschungsbereich „Bausteine der Materie und fundamentale Wechselwirkungen“ der Universität Bonn unterstützt, dessen Mitglied Prof. Schiemann ist.
Publikation: Tobias Hett et al.: Spatio-Temporal Resolution of Conformational Changes in Biomolecules by Combining Pulsed Electron-Electron Double Resonance Spectroscopy with Microsecond Freeze-Hyperquenching; Journal of the American Chemical Society; DOI: https://doi.org/10.1021/jacs.1c01081
Kontakt:
Prof. Dr. Ulrich Benjamin Kaupp
Emeritus Direktor, Forschungszentrum caesar
LIMES-Institut
Tel. 0228/9656100
E-Mail: U.B.Kaupp@caesar.de
Prof. Dr. Olav Schiemann
Institut für Physikalische und Theoretische Chemie
Universität Bonn
Tel. 0228/732989
E-Mail: schiemann@pc.uni-bonn.de