Technik-Plattformen
- Hochauflösende Lichtmikroskopie
- Genetic engineering facility
- Massenspektrometrie
- Konfokale Laser Scanning Mikroskopie
- Life Cell Imaging
- PRECISE Platform foR SinglE Cell GenomIcS and Epigenomics
- Selektion von Aptameren in der Plattform CARD
Hochauflösende Lichtmikroskopie

In den Lebenswissenschaften wird die Lichtmikroskopie weit verbreitet eingesetzt, um zelluläre Strukturen zu visualisieren. Allerdings limitiert die Beugung die Auflösung von Lichtmikroskopen auf 250 nm. Diese Auflösungsgrenze ist nicht ausreichend, um zelluläre Details wie dicht zusammenliegende Filamente, Anhäufungen von kleinen Objekten oder Organell-umhüllende Membranen aufgelöst darzustellen.
STED (stimulated emission depletion)-Mikroskopie nutzt bestimmte Eigenschaften von fluoreszierenden Farbstoffen, um die beugungslimitierte Auflösungsgrenze zu umgehen. Die Methode wurde in den 90ern erfunden und realisiert und zu Beginn des Jahrtausends weiter verbessert und kommerziell verfügbar gemacht. Für seine Arbeiten zur STED-Mikroskopie wurde Stefan Hell mit dem Nobelpreis für Chemie 2014 ausgezeichnet.
Seit Sommer 2015 gibt es in der Einrichtung für hochauflösende Mikroskopie am LIMES-Institut ein "4-channel easy3D superresolution STED microscope (Abberior Instruments)", das Aufnahmen mit einer Auflösung bis zu 30 nm ermöglicht. Im Gegensatz zur Elektronenmikroskopie, die traditionell eingesetzt wird, um zelluläre Details mit hoher Auflösung zu visualisieren, erfordert die Methode nicht das Einbetten der Probe, das Schneiden der Probe in dünne Schnitte und die Aufnahme im Vakuum. Es können sogar lebende Zellen untersucht werden. Weiterhin kann die spezifische Markierung von Proteinen leichter erreicht werden als in der Elektronenmikroskopie.
Zellen sind die kleinsten Einheiten des Lebens. Ihre Architektur und ihre Komponenten zu verstehen ist entscheidend für die Weiterentwicklung der Lebenswissenschaften. Die STED-Mikroskopie erlaubt das Studium verschiedenster zellulärer Details wie Nanodomänen, Organellen oder dem Zytoskelett und trägt daher dazu bei, die molekulare Komplexität von lebender Materie zu enträtseln.
Anprechpartner: Prof. Dr. Thorsten Lang, thorsten.lang@uni-bonn.de
Genome Editing Core Facility

Die Entwicklung von CRISPR/Cas9 im letzten Jahrzehnt hat die gezielte Manipulation der Erbinformation in embryonalen Stammzellen und in befruchteten Eizellen enorm vorangebracht. Damit wurde den Ablauf solcher Projekte stark beschleunigt und auch komplexere Veränderungen ermöglicht. Am LIMES-Institut werden diese Techniken in Drosophila melanogaster, Xenopus tropicalis sowie in Zebrafisch- und Mausmodellen vielseitig eingesetzt. Die Genome Editing Core Facility ist dabei Ihr Ansprechpartner von der Beratung bis zur Durchführung ganzer Projekte.
Ansprechpartner: Dr. Stephan Sonntag, stephan.sonntag@uni-bonn.de
Massenspektrometrie
Massenspektrometrie ist die wichtigste analytische Technologie zur Identifizierung und Quantifizierung von Proteinen, Lipiden, Kohlenhydraten, kleinen Metaboliten und pharmakologisch aktiven Substanzen. LIMES besitzt ein Massenspektrometer neuester Bauart, ein Thermo Q Exactive Plus, das hauptsächlich für Lipidomik und Metabolomik eingesetzt wird. Es ist von großer Bedeutung für Forschung am Lipidstoffwechsel, Stoffwechselerkrankungen und deren Beziehung zum Immunsystem. Die Präzision der Messungen von typischerweise 0,001 Da ermöglicht die Identifizierung von Substanzen nur durch ihre Masse; die extrem hohe Auflösung von 280000 ermöglicht die Trennung von Massenpeaks, die sich um nicht mehr als 0,002 Da unterscheiden.
Ansprechpartner: Prof. Dr. Christoph Thiele, cthiele@uni-bonn.de
Konfokale Laser Scanning Mikroskopie

Kernstück der Plattform sind zahlreiche hochmoderne Systeme für die konfokale Visualisierung von Zellen und Geweben. Darüberhinaus unterstützen wir mit unserer wissenschaftlichen und technischen Expertise Nutzer bei der Planung, Durchführung und Analyse ihrer Experimente und der Mikroskopie. Die Plattform ist mit zahlreichen Standard- und hochentwickelten Spezialsystemen ausgestattet, die sowohl hochauflösende als auch ultra-schnelle konfokale Bildgebung ermöglichen. Kolokalisierungsstudien können ebenso durchgeführt werden wie FRAP (fluorescent recovery after photobleaching), FRET (fluorescence resonance energy transfer) und die Visualisierung von photoaktivierbaren Proteinen. Für die Bildanalyse in 2D, 3D und 4D stehen spezielle Softwareprogramme und leistungsfähige Workstations zur Verfügung.
Anprechpartner: Dr. Thomas Quast, quast@uni-bonn.de
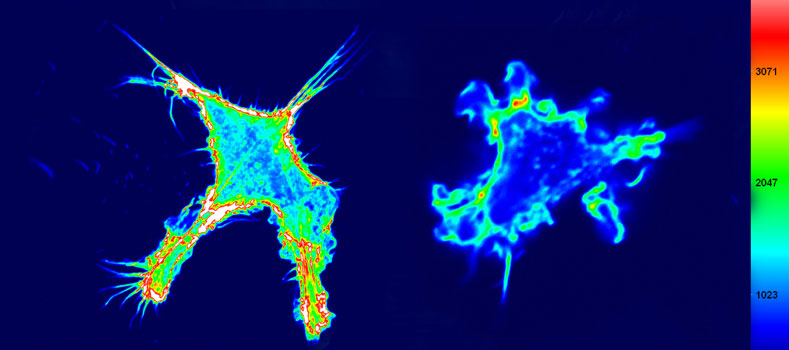
Life Cell Imaging
Die "Live Cell Imaging"-Plattform bietet spezialisierte mikroskopische Systeme für die Videomikroskopie von intrazellulären dynamischen Prozessen und Zellmotilität. Wir verfügen über langjährige Expertise in dem Gebiet der Adhäsion, Migration und Chemotaxis von Zellen. Zur Analyse von Zellbewegung mit hoher räumlicher und zeitlicher Auflösung haben wir zahlreiche mikroskopische Untersuchungsmethoden entwickelt. Kürzlich haben wir aufwendige Mikrofluidik-Techniken etabliert, mit denen wir hochaufgelöst direktionale Migration von Immunzellen in präzise kontrollierbaren Chemokingradienten untersuchen können. Technisch ist die Plattform mit diversen automatisierten video-mikroskopischen Systemen ausgestattet, einschließlich Durchlicht-Kontrastverfahren, Weitfeld-Epifluoreszenzdetektion und konfokalen Mikroskopen. Schließlich bieten wir für die Nutzer qualifizierte wissenschaftliche und technische Unterstützung sowohl bei der Planung und Durchführung der Experimente als auch beim Training an den Systemen.
Ansprechpartner: Dr. Thomas Quast, quast@uni-bonn.de
PRECISE Platform foR SinglE Cell GenomIcS and Epigenomics

Mit der Entwicklung der Einzelzell-RNA-Sequenzierung wurde das Forschungsgebiet der Transkriptom-Analyse und Systembiologie revolutioniert. Während mit bisherigen Methoden, wie etwa FACS, die Identifizierung einzelner Zellen lediglich auf einer limitierten Anzahl vordefinierter Marker beruhte, können mit Hilfe der Einzelzell-RNA-Sequenzierung Zellen anhand mehrerer hundert bis tausend Gene charakterisiert werden. So führte die Anwendung dieser Technologie beispielsweise zur Entdeckung bisher unbekannter Neuronen (Zeisel et al., Science, 2015) und Immunzellen (Villani et al., Science, 2017).
Um den Einzug der Einzelzell-Technologien in den Laboralltag weiter voranzutreiben, wurde die "Platform for Single Cell Genomics and Epigenomics" ins Leben gerufen. PRECISE ist ein Joint Venture zwischen der Universität Bonn und dem Deutschen Zentrum für Neurodegenerative Erkrankungen (DZNE).
Dabei ist ein besonderer Schwerpunkt, genomische Technologien auf Einzelzell-Ebene zu entwickeln, anzuwenden und internen wie externen Kooperationspartnern verfügbar zu machen.
Im Rahmen des PRECISE werden folgende Einzelzell-Techniken verwendet:
- MARS-Seq: MARS-Seq (Jaitin et al., Science, 2014) ist eine automatisierte hoch-parallelisierte Methode der Einzellzell-Sequenzierung. Die Methode beruht auf der Isolierung einzelner Zellen mit Hilfe von FACS mit anschließender RNA-Sequenzierung. Da die Informationen des FACS gespeichert werden, können diese später mit der Transkriptom-Information verknüpft werden.
- SMART-Seq2: Die SMART-Seq2 Methode (Picelli et al., Nature Protocols, 2013) verwendet die SMART-Reaktion (Switching Mechanism At the end of the 5′-end of the RNA Transcript). Der Vorläufer dieser Methode wurde bereits 2013 entwickelt (Picelli et al., Nature Methods, 2013). Während MARS-Seq nur das 3‘-Ende sequenziert, ermöglicht SMART-Seq2 die Sequenzierung des gesamten Transkripts.
- Drop-Seq: Da Einzelzell-Technologien häufig besser und reproduzierbarer in Nanoliter-Reaktionen ablaufen, wurde Drop-Seq (Macosko et al., Cell, 2015) entwickelt, welches sich die Mikrofluidik zu Nutze macht. Mit Drop-Seq können in kurzer Zeit und geringem Kostenaufwand Tausende von Einzelzellen sequenziert werden.
- SeqWell: Eine Alternative zu Drop-Seq bietet das SeqWell-System (Gierahn et al., Nature Methods, 2017). Auch hier werden die Reaktionen im Nanoliter-Volumen durchgeführt. Im Gegensatz zu Drop-Seq erfolgt dies aber in Mikrowells, kombiniert mit einer semi-permeablen Membran. Ein großer Vorteil des SeqWells ist die Portabilität dieser Technologie.
- C1: Die C1-Plattform beruht, ähnlich wie Drop-Seq, auf Mikrofluidik und wird von Fluidigm vertrieben. Ein besonderes Feature dieser Technologie ist das Festsetzen der Einzelzellen in spezifischen "capture sites", wo sie visualisiert werden können. PRECISE benutzt C1 um SMART-Seq durchzuführen.
Anprechpartner: Prof. Dr. Joachim L. Schultze, Office.immunogenomics@uni-bonn.de
Selektion von Aptameren in der Plattform CARD

Aptamere sind kurze einzelsträngige Nukleinsäuren, die aufgrund ihrer 3D-Struktur in der Lage sind ähnlich wie Antikörper hochaffin und hochspezifisch an verschiedenste Zielmoleküle zu binden. Die Identifizierung von Aptameren erfolgt mittels einer in vitro Selektions-Methode (SELEX-Methode) nach dem Kriterium einer hohen Bindungsaffinität. Mittels dieser Methode lassen sich Aptamere gegen Toxine, Peptide, Proteine, Viren, Bakterien und sogar gegen ganze Zellen selektieren. Durch den Einbau von modifizierten Nukleotiden können spezifische Aptamere, die gegen den Abbau durch Nukleasen resistent sind, zu analytischen oder therapeutischen Zwecken entwickelt und auch für die Sichtbarmachung von Strukturen verwendet werden. Die vielseitigen Einsatzmöglichkeiten von Aptameren machen sie zu universellen Biomolekülen und zu einer zuverlässigen Alternative zu Antikörpern.
Seit Januar 2017 besteht an der Universität Bonn das „Center of Aptamer Research and Development“ (CARD), ein Geräte Zentrum welches Serviceleistungen im Rahmen der Selektion von Aptameren für akademische Gruppen aber auch für Firmen zur Verfügung stellt. Folgende Leistungen werden angeboten: Die Generierung von neuen Aptameren mittels eines automatisierten in vitro Selektionsverfahren, regelmäßigen Workshops im Rahmen der Aptamer Selektion sowie die praktische und theoretische Unterstützung im Bereich der Aptamer-Forschung. Für weitere Informationen besuchen Sie bitte unsere Website www.aptamer-center.de.
Kontakt:
Prof. Dr. Günter Mayer, gmayer@uni-bonn.de